こんにちは! 質量分析屋の髙橋です。プロテオミクスの研究者の中には、LC/MS/MSのData Dependent Acquisition(DDA)の機能を使ってプロダクトイオンを取得し、タンパク質の同定を行っている方が沢山いらっしゃると思います。分子量にも依りますが、ペプチドの多くはESIにおいて多価イオンを生成するために、DDAの設定で“多価イオンのみをプリカーサーイオンとして選択する”機能を使う場合が殆どです。
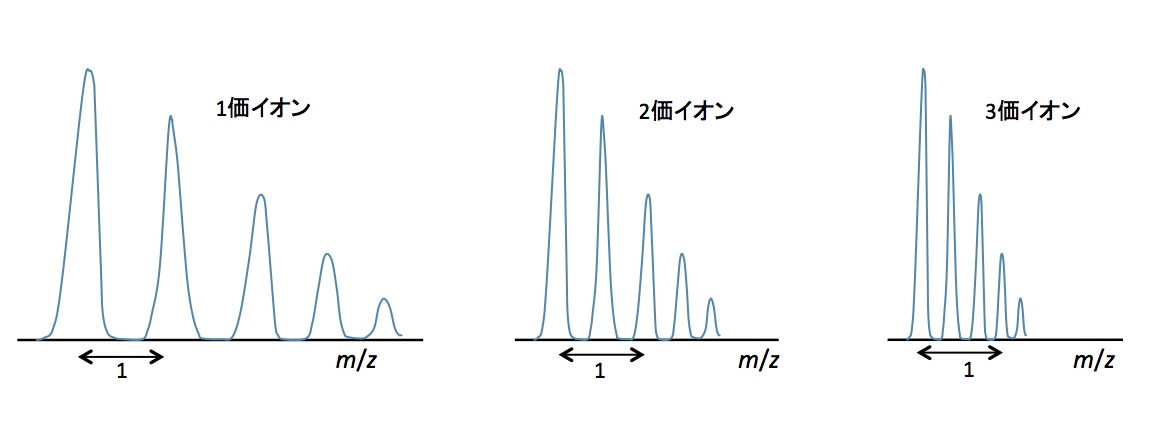
この機能は、高分解能質量分析計で用いられる場合が多く、多価イオンであるか否かをシステムが認識するのは、同位体ピークの分離挙動だと推測されます。即ち、図1に示すように同位体ピークのm/z間隔が、1価イオンは1、2価イオンは1/2、3価イオンは1/3になる事に依るものです。
図1 イオンの価数と同位体分離挙動
ここで、ESIでは試料成分の濃度が高い時、クラスターイオンが生成される事が知られています。そして問題になるのが、クラスターイオンの多価イオンも生成される事があると言うことです。
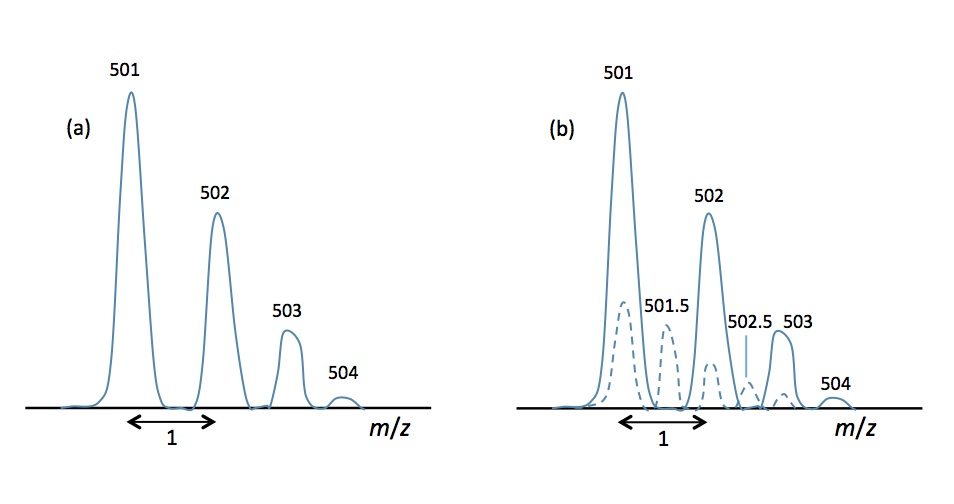
例えばペプチド混合物の中に、ノミナル質量500と言う低分子化合物が大量に含まれていて、イオン化されたとします。ここで、図2(a)に示す様にプロトン付加分子のみが観測されれば、全く問題はありません。しかし、図2(b)のように、2量体の2価イオンが1価イオンに重なって観測されると、システムはこのイオンをノミナル質量1,000の化合物の2価イオンであると誤認識してしまい、プロダクトイオンスペクトルを測定してしまいます。
図2(a)1価イオンの同位体分離パターン、(b)二量体の2価イオンが混ざったパターン
通常の低分子化合物の2量体の2価イオンからのプロダクトイオンスペクトルは、ペプチドとは似ても似つかないパターンになると思われるので、タンパク質データベースで検索しても何もヒットしてこないとは思いますが、排除するに越したことはないですね。同位体ピークの強度比を見れば一目瞭然なので、生データを見れば容易に排除できます。ソフトに100%頼る事なく、生データを見る癖をつける事をお勧めします。



